Prof. Dr. Tobias Madl
Ordnung in die Protein Unordnung!

Lehrstuhl für Molekularbiologie und Biochemie
BioTechMed -Universität Graz
Mozartgasse 12/II
8010 Graz
Email:tobias.madl@medunigraz.at
Forschungsgebiet
“Funktion benötigt Struktur” – das ist eine vorherrschende Meinung in der Strukturbiologie. In der Tat benötigen die meisten Proteine eine definierte 3-dimensionale Struktur um ihre Funktion auszuüben. Ein substantieller Anteil des Genoms jedes Organismus kodiert jedoch Proteine die, entweder vollständig oder zum Teil, keine definierte 3-dimensionale Struktur benötigen um ihre Funktion auszuüben, aber essentiell für zelluläre Funktionen sind: die sogenannten intrinsisch unstrukturierten Proteine/Regionen (intrinsically oder natively disordered proteins/regions (IDP/Rs)). Aufgrund des häufigen Auftretens in Krankheiten gibt es keinen Zweifel dass diese Proteine bevorzugte Angriffspunkte in der Entwicklung von Wirkstoffen sind. Um diese Proteine generell zu verstehen, und um Strategien zu entwickeln um deren Interaktionen für die Behandlung von Krankheiten zu modulieren, ist die Erkennung struktureller Eigenschaften sowie die detaillierte Charakterisierung der biologischen Komplexe der Schlüssel.
Meine BioSysNet-finanzierte Forschung konzentriert sich auf die Schlüssel-IDP/Rs im Schnittpunkt der Wnt und Insulin Signalwege welche grundlegende Entscheidungen über das Zellschicksal determinieren, sowie zu Krankheiten und Alterung führen. Kürzlich konnte gezeigt werden dass die Regulation dieser Proteine einen komplizierten Code post-translationaler Modifikationen (post-translational modifications; PTMs) und die Bindung einer Fülle an Ko-Faktoren (z.B. ß-catenin) innerhalb deren IDRs umfasst. Störungen dieser komplizierten Protein-Protein Interaktionsnetzwerke durch Mutationen sowie Änderungen im Status der Signalwege (unterschiedliche PTMs) führen zu Krebs, Diabetes und Alterung. Wenngleich dieses System von immenser Wichtigkeit ist, sind die grundlegenden molekularen Mechanismen weitgehend unbekannt. Dies liegt hauptsächlich an dessen intrinsischer Flexibilität, was die strukturelle Charakterisierung durch herkömmliche Ansätze erschwert.
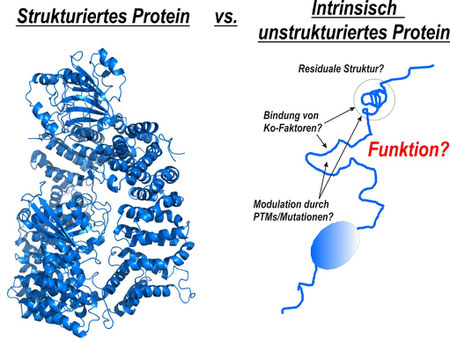
Um dieses Problem in Angriff zu nehmen, verwende ich meinen kürzlich entwickelten multidisziplinären strukturbiologischen Ansatz um die strukturellen Eigenschaften Krankheits-relevanter IDRs alleine und in Komplex mit deren biologischen Ko-Faktoren zu charakterisieren, sowie die Modulation residualer Struktur durch Modifikationen (PTMs, Mutationen) und die Wechselwirkungen kleiner organischer Wirkstoffe mit deren Funktionen in vitro und in vivo zu untersuchen. Dies liefert den Schlüssel zum Verständnis dieser Proteine im Allgemeinen und vermittelt Einblick wie deren Interaktionen zur Behandlung von Krankheiten moduliert werden können.
Mitarbeiter
Dr. Christoph Göbl
Christoph Hartlmüller
Dr. Henry Hocking
Dr. Benjamin Bourgeois
Dr. Sandra Scanu
Publikationen im Rahmen von BioSysNet
Huang JR, Warner LR, Sanchez C, Gabel F, Madl T, Mackereth CD, Sattler M, Blackledge M (2014). Transient electrostatic interactions dominate the conformational equilibrium sampled by multidomain splicing factor U2AF65: a combined NMR and SAXS study. J Am Chem Soc 136(19):7068-76.
Lorenz OR, Freiburger L, Rutz DA, Krause M, Zierer BK, Alvira S, Cuéllar J, Valpuesta JM, Madl T, Sattler M, Buchner J (2014). Modulation of the Hsp90 chaperone cycle by a stringent client protein. Mol Cell 53(6):941-53.
Göbl C, Madl T, Simon B, Sattler M (2014). NMR approaches for structural analysis of multidomain proteins and complexes in solution. Prog Nucl Magn Reson Spectrosc 80:26-63.
Müller R, Gräwert MA, Kern T, Madl T, Peschek J, Sattler M, Groll M, Buchner J (2013). High-resolution structures of the IgM Fc domains reveal principles of its hexamer formation. High-resolution structures of the IgM Fc domains reveal principles of its hexamer formation. Proc Natl Acad Sci U S A 110(25):10183-8.
Putker M, Madl T, Vos HR, de Ruiter H, Visscher M, van den Berg MC, Kaplan M, Korswagen HC, Boelens R, Vermeulen M, Burgering BM, Dansen TB (2013). Redox-dependent control of FOXO/DAF-16 by transportin-1. Mol Cell 49(4):730-42.
Dormann D, Madl T, Valori CF, Bentmann E, Tahirovic S, Abou-Ajram C, Kremmer E, Ansorge O, Mackenzie IR, Neumann M, Haass C (2012). Arginine methylation next to the PY-NLS modulates Transportin binding and nuclear import of FUS. EMBO J 31(22):4258-75.
Hocking HG, Zangger K, Madl T (2013). Studying the structure and dynamics of biomolecules by using soluble paramagnetic probes. Chemphyschem 14(13):3082-94.
Karagöz GE, Duarte AM, Akoury E, Ippel H, Biernat J, Morán Luengo T, Radli M, Didenko T, Nordhues BA, Veprintsev DB, Dickey CA, Mandelkow E, Zweckstetter M, Boelens R, Madl T, Rüdiger SG (2014). Hsp90-Tau Complex Reveals Molecular Basis for Specificity in Chaperone Action. Cell 156 (5):963-974.
Zhang Y, Madl T, Bagdiul I, Kern T, Kang HS, Zou P, Mäusbacher N, Sieber SA, Krämer A, Sattler M (2013). Structure, phosphorylation and U2AF65 binding of the N-terminal domain of splicing factor 1 during 3'-splice site recognition. Nucleic Acids Res 41(2):1343-54.
Publikationen vor BioSysNet
Dormann D., Madl T., Valori C.F., Bentmann E., Tahirovic S., Abou-Ajram C., Kremmer E., Ansorge O., Machenzie I.R.A., Neumann M.; Haass C., Arginine methylation next to PY-NLS modulates Transportin binding and nuclear import of FUS, EMBO (2012), 1-18
Tavanez J.P., Madl T., Kooshapur H., Sattler M., Valcárcel J., hnRNP A1 proofreads 3’ splice site recognition by U2AF, Molecular Cell (2012)
Tripsianes K., Madl T., Machyna M., Fessas D., Englbrecht C., Fischer U., Neugebauer K.M., Sattler M., Structural basis for dimethyl-arginine recognition by the Tudor domains of SMN and SPF30,
Nature Structural and Molecular Biology 18 (2011), 1414-1420
Mackereth C.D., Madl T., Bonnal S., Simon B., Zanier K., Gasch A., Rybin V., Valcárcel J. and Sattler M., Multi-domain conformational selection underlies polypyrimidine recognition by U2AF and
pre-mRNA splicing, Nature 475 (2011), 408-411
Madl T., Güttler T., Görlich D., Sattler M., Structural Analysis of Large Protein Complexes using Solvent Paramagnetic Relaxation Enhancements, Angewandte Chemie 50 (2011), 3993-3997
Madl T., Gabel F., Sattler M., NMR and Small Angle Scattering-based structural analysis of protein complexes in solution, Journal of Structural Biology 173 (2011), 472-482
Güttler T., Madl T. (shared first author), Neumann P., Deichsel D., Corsini L., Monecke T., Ficner R., Sattler M., Görlich D., Structural basis for the recognition of diverse nuclear export
signals by the exportin CRM1, Nature Structural and Molecular Biology 17 (2010), 1367-1376
Meyer H., Tripsianes K., Vincendeau M., Madl T., Kateb F., Brack-Werner R., Sattler M., Structural basis for homodimerization of the Src-associated during mitosis, 68 kD protein (Sam68) Qua1
domain, Journal of Biological Chemistry 285 (2010), 28893-28901
Madl T., Felli I.C., Bertini I., Sattler M., Structural analysis of protein interfaces from 13C direct-detected paramagnetic relaxation enhancements, Journal of the American Chemical Society 132
(2010), 7285-7287
Simon B., Madl T., Mackereth C.D., Nilges M., Sattler M., An Efficient Protocol for NMR-Spectroscopy-Based Structure Determination of Protein Complexes in Solution, Angewandte Chemie 49 (2010),
1967-1970
Spirk S., Belaj F., Madl T., Pietschnig R., A Radical Approach to Hydroxylaminotrichlorosilanes: Synthesis, Reactivity, and Crystal Structure of TEMPO-SiCl3 (TEMPO =
2,2,6,6-Tetramethylpiperidine-N-oxyl), European Journal of Inorganic Chemistry 2 (2010), 289-297
Madl T., Bermel W., Zangger K.: Use of Relaxation Enhancements in a Paramagnetic Environment for the Structure Determination of Proteins Using NMR Spectroscopy, Angewandte Chemie 48 (2009),
8259-8262
Madl T., Sattler M.: Adhesion dance with raver, Structure 17 (2009), 781-783
Zangger K., Respondek M., Göbl C., Hohlweg W., Rasmussen K., Grampp G., Madl T.: Positioning of micelle-bound peptides by paramagnetic relaxation enhancements, Journal of Physical Chemistry B 113
(2009) 4400-4406
Traar P., Rumpler A., Madl T., Saischek G., Francesconi KA.: Synthesis of Naturally Occurring Arsenic-Containing Carbohydrates, Australian Journal of Chemistry 6 (2009), 538-545
Neufeld C., Filipp FV., Simon B., Neuhaus A., Schüller N., David C., Kooshapur H., Madl T., Erdmann R., Schliebs W., Wilmanns M., Sattler M..: Structural basis for competitive interactions of
Pex14 with the import receptors Pex5 and Pex19, EMBO Journal 28 (2009) 745-754
Spirk S., Madl T., Pietschnigg R., Relax with TEMPO: A Paramagnetic Relaxation Agent Useful also for Silicon-29 NMR Spectroscopy, Organometallics 27 (2009), 500-502
Respondek M., Madl T., Gobl C., Golser R., Zangger K., Mapping the Orientation of Helices in Micelle-Bound Peptides by Paramagnetic Relaxation Waves, Journal of the American Chemical Society 129
(2007), 5228-5234
Madl T., Van Melderen L., Mine N., Respondek M., Oberer M., Keller W., Khatai L., Zangger K., Structural basis for nucleic acid and toxin recognition of the bacterial antitoxin CcdA, Journal of
Molecular Biology 364 (2006), 170-185
Madl T., Sterk H., Rechberger G.N., Mittelbach M., Tandem mass spectrometric analysis of a complex triterpene saponin mixture of Chenopodium quinoa, Journal of the American Society for Mass
Spectrometry 17 (2006), 795-806
Madl T., Mittelbach M., Quantification of primary fatty acid amides in commercial tallow and tallow fatty acid methyl esters by HPLC-APCI-MS, The Analyst 130 (2005), 565-570



